Rekombinacja jest to proces wymiany materiału genetycznego, w wyniku którego powstają nowe genotypy. Stanowi ona główne źródło występującej w przyrodzie zmienności dziedzicznej oraz podstawę dla procesów ewolucyjnych. Jest również jedną z metod usuwania uszkodzeń powstałych w DNA. Nie prowadzi ona jednak do powstawania nowych genów, lecz jedynie do tworzenia się różnych kombinacji uprzednio istniejących sekwencji.
Rekombinacja u organizmów wyższych
U organizmów rozmnażających się płciowo rekombinacja jest wynikiem losowego łączenia się gamet o różnym składzie genetycznym, a także segregacji chromosomów i procesu crossing- over zachodzącego w trakcie mejozy.
Równa i wzajemna wymiana informacji genetycznej wymaga precyzyjnego, równoległego ustawienia się chromosomów homologicznych. W rzadkich przypadkach ich ułożenie nie jest dokładne i zdarzenia rekombinacyjne powodują nierówną wymianę informacji. Jeden z chromosomów otrzymuje wówczas nadmiar materiału genetycznego, natomiast drugi zaopatrzony jest w niekompletną informację (delecja). Tzw. nierówny crossing-over oddziałuje na tandemową organizację DNA.
Dzięki odmiennemu połączeniu i współdziałaniu genów nieallelicznych, gamety różnią się od siebie genotypem, a wśród potomstwa nie pojawiają się identyczne osobniki. Odróżniają się one także od form rodzicielskich. Umożliwia to rozmnażającym się płciowo organizmom uniknięcie zbytniego nagromadzenia się szkodliwych mutacji, czyli tzw. zapadki Mullera.
Regulacja poziomu rekombinacji u różnych organizmów musi podlegać selekcji. Zbyt niski poziom rekombinacji uniemożliwiałby genetyczną adaptację osobników do zmiennych warunków środowiskowych. Zbyt wysoki wpłynąłby natomiast na zbyt szybkie rozwijanie się kombinacji genowych, wyselekcjonowanych w konkretnych warunkach bytowych danego gatunku.

Rys.1 Crossing-over czyli wymiana materiału genetycznego między chromosomami homologicznymi
Rekombinacja u bakterii
U bakterii rekombinacja towarzyszy procesom transdukcji (przenoszenie materiału dziedzicznego bakterii za pośrednictwem bakteriofagów) i transformacji (wymiana materiału genetycznego między komórkami bakteryjnymi, która może powodować m.in. uzjadliwianie się niektórych szczepów).
Typy rekombinacji DNA
Wyróżnia się cztery podstawowe typy rekombinacji genetycznej.
1) Rekombinacja homologiczna lub inaczej uprawniona, uogólniona – zachodzi między cząsteczkami DNA o wysokiej homologii, np. wewnątrz genomu diploidalnego organizmu. Wymaga ona obecności odpowiednich enzymów lub szlaków enzymatycznych umożliwiających kolejno zerwanie i połączenie przylegających do siebie homologicznych fragmentów DNA. Ważną rolę w jej przebiegu odgrywa białko RecA, aktywowane przez ATP. Ułatwia ono jednoniciowemu DNA szybkie przeszukiwanie dupleksu w celu znalezienia identycznych lub podobnych sekwencji, a następnie katalizuje wymianę nici w miejscu największej homologii.
Ten typ rekombinacji odgrywa kluczową rolę przy naprawie materiału genetycznego. W przypadku, gdy obie nici dupleksu są uszkodzone, informacja niezbędna do ich naprawy musi pochodzić z innej cząsteczki DNA. Rekombinacja dostarcza zatem matrycy dla syntezy odpowiedniego łańcucha wypełniającego ubytek. DNA mutantów niezdolnych do rekombinacji jest narażony na nieodwracane uszkodzenia spowodowane działaniem promieniowania UV oraz innych czynników mutagennych.
Istnieją trzy różne modele rekombinacji homologicznej:
• Najstarszym z nich jest model Robina Hollidaya zwany również rekombinacją ogólną. Ustanowiony został w 1964 r. na podstawie produktów mejozy u grzybów i stanowił podstawę do dalszych badań nad mechanizmami rekombinacji.
W początkowym etapie, dwa homologiczne dupleksy ustawiają się względem siebie w odpowiedni sposób. W każdym z dupleksów jeden łańcuch jest przecinany przez enzym endonukleazę. Powstałe wolne końce opuszczają swój wyjściowy dupleks i łączą się z homologicznymi fragmentami drugiego dupleksu DNA, tworząc w ten sposób dynamiczny intermediat rekombinacyjny. Następnie zachodzi proces przemieszczania się ramion (ang. branch migration) i przesunięcie punktu krzyżowania się nici (ang. crossover). Powstały produkt pośredni tzw. figura krzyżowa Robina Hollidaya może być przycinany i ponownie łączony na dwa sposoby.
Przecięcie nici niedokonujących inwazji, prowadzi do powstania produktów, których jedna połowa pochodzi z jednego rodzicielskiego dupleksu DNA, a druga z drugiego. Jeżeli przecięciu ulegną dwie, dokonujące inwazji nici, tworzą się cząsteczki różniące się od rodzicielskich jedynie wymienionymi regionami.
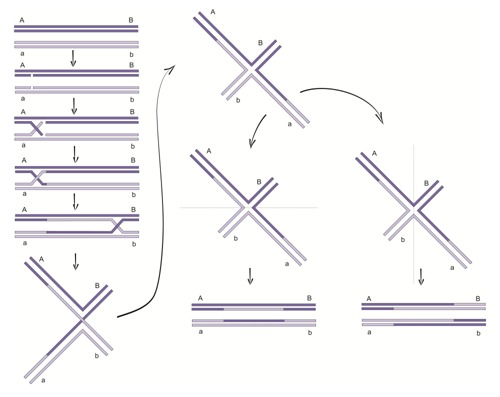
Rys.2 Model Hollidaya rekombinacji homologicznej
• model Meselsona-Raddinga lub inaczej model Aviemore
Ten typ rekombinacji rozpoczyna się od nacięcia pojedynczej nici jednego z homologów, po którym następuje synteza DNA z końca 3’ odsuwająca koniec 5’ przerwanej nici. Dokonuje on inwazji nici homologicznej i odsuwa swój nukleolitycznie degradowany odpowiednik tworząc przy tym pętlę D (odsunięcie jednej nici z dwuniciowego DNA podczas rekombinacji). Następująca potem ligacja prowadzi do wytworzenia genetycznie niesymetrycznego połączenia Hollidaya, w którym tylko jedna z podwójnych nici zawiera region heterodupleksowy. Może dojść również do migracji połączenia, po którym heterodupleksy tworzą się na obu niciach. Rozdzielenie połączenia może zachodzić na dwa sposoby, tak jak ma to miejsce w modelu Hollidaya.

Rys.3 Model Meselsona-Raddinga rekombinacji homologicznej
• model Szostaka – model przerywania obu nici
Model Szostaka rozpoczyna się dwuniciowym pęknięciem (ang. double strand break – DSB) jednego z homologów. Następnie w wyniku działalności egzonukleazy 5’ -> 3’ powstają 3’ wystające końce, z których jeden dokonuje inwazji homologicznego dupleksu, a następnie odsuwa swój odpowiednik wytwarzając pętlę D. Do końców 3’ dołącza się polimeraza DNA, która prowadzi syntezę łańcucha, a ligaza odtwarza dwuniciowe struktury, tworząc w ten sposób połączenia Hollidaya.
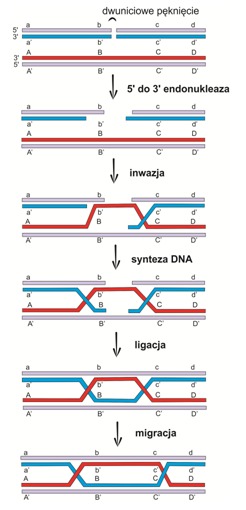
Rys.4 Model Szostaka rekombinacji homologicznej
2) Konwersja genów – na skutek naprawy uszkodzeń DNA jeden z alleli przekształcany jest w drugi. Proces ten, nazywany też korektą sekwencji, może zachodzić wewnątrz jednego chromosomu lub pomiędzy dwoma chromosomami.
3) Rekombinacja zlokalizowana, umiejscowiona lub specyficzna dla miejsca (ang. site-specific) – wymaga jedynie krótkich obszarów homologii.
Przykładowo zachodzi podczas integracji faga λ, którego kolista cząsteczka DNA jest zdolna do ekspresji i replikacji dzięki współdziałaniu z białkami gospodarza. Rekombinacja zlokalizowana odpowiedzialna za włączenie faga do chromosomu bakteryjnego katalizowana jest przez białka rozpoznające specyficzne sekwencje DNA, nie zaś sekwencje identyczne lub podobne, tak jak w przypadku rekombinacji homologicznej. W tym procesie nie uczestniczy również białko RecA, a do jej zapoczątkowania nie jest potrzebny jednoniciowy DNA.
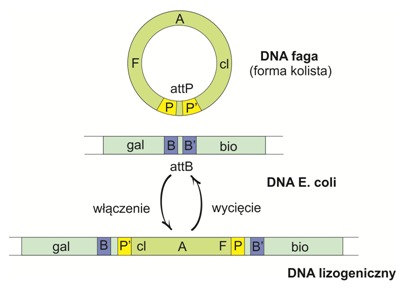
Rys.5 Schemat rekombinacji DNA faga λ oraz genomu E.coli. DNA λ
Tego typu rekombinacja występuje również podczas przełączania typu koniugacyjnego u drożdży, tworzenia się spor u Bacillus subtilis oraz różnicowania się heterocyst u sinic Klebsiella i Anabaena.
4) Rekombinacja niehomologiczna, inaczej nieuprawniona, transpozycja – zachodzi między niespokrewnionymi sekwencjami, np. przemieszczenia się fragmentów chromatyd w obrębie chromosomów niehomologicznych.
Do jej realizacji konieczna jest obecność specyficznych enzymów rozpoznających sygnalne sekwencje w obu, ulegających rekombinacji cząsteczkach.
Autor: Anna Kurcek
Literatura:
1. Stryer L. 2003. Biochemia. Wydawnictwo Naukowe PWN, Warszawa.
2. Murray R. P., Granner D. K., Mayes P. A., Rodwell V. W., 1995. Biochemia Harpera. Wydawnictwo Lekarskie PZWL, Warszawa.
3. Sadakierska-Chudy A., Dąbrowska G., Goc A. 2004. Genetyka ogólna. Skrypt do ćwiczeń dla studentów biologii. Uniwersytet Mikołaja Kopernika, Toruń.
4. Ross C. H., 2005. Working with Molecular Genetics. Chapter 8 Recombination of DNA. The Pennsylvania State University.

