Antyczny DNA (aDNA) to materiał genetyczny pochodzący z organizmów dawno żyjących pozyskiwany z wielu różnych źródeł w tym z kości, wytworów skóry, szczątków zmineralizowanych ale także osadów, gleby i lodowców w przypadku antycznego DNA środowiskowego. Kwasy nukleinowe, pomimo podatności na czynniki środowiskowe zarówno chemiczne jak i fizyczne, jeżeli znajdą się w odpowiednich warunkach są cząsteczkami trwałymi. DNA natomiast, z uwagi na swoją najczęściej dwuniciową postać, jest bardziej odporne na uszkodzenia i czynniki modyfikujące niż RNA.
Przełomem w naszej wiedzy o tym jak długo cząsteczki DNA mogą się opierać degradacji post mortem na tyle, aby wciąż można było je wyizolować, namnożyć, zsekwencjonować i poddać dalszym analizom był rok 1984, w którym dr Russel Higuchi z Uniwersytetu Kalifornijskiego wraz z zespołem zsekwencjonowali mtDNA wyizolowany z zakonserwowanych przez wysuszenie mięśni Zebry kwagga (Equus quagga quagga martwej od około 140 lat. Był to pierwszy przypadek kiedy udało się zsekwencjonować DNA pochodzący od wymarłego gatunku. Co więcej, po udanym odczytaniu sekwencji mtDNA z pobranej próbki udało się ją przyrównać do sekwencji DNA obecnie żyjącej Zebry górskiej (Equus zebra) i ustalić, że różnią się między sobą w dwunastu pozycjach a dalsze analizy filogenetyczne pozwoliły oszacować, że wspólny przodek tych dwóch gatunków żył 3-4 miliony lat temu.
Praca dr Higuchi’ego wykazała, że analiza materiału genetycznego z tego typu próbek jest możliwa i zapoczątkowała rozwój badań nad antycznym DNA, które wraz z upływem czasu dostarczały coraz więcej informacji i obejmowały nowe gałęzie nauk biologicznych, w tym obszar badań dotyczący migracji populacji ludzkich czy analiza porównawcza materiału genetycznego wczesnych przedstawicieli Homo sapiens, Neandertalczyków i Denisowian. Badania te dostarczyły dane pozwalające nam przypuszczać, że wczesny Homo sapiens krzyżował się ze wspomnianymi innymi homininami. Należy jednak pamiętać, że nie dysponujemy wystarczającą ilością danych aby uznać występowanie krzyżówek pomiędzy tymi trzema grupami homininów za wysoce prawdopodobne i powszechne. Obecny stan wiedzy kieruje nas ku stwierdzeniu, że jeżeli związki seksualne pomiędzy H.sapiens, Neandertalczykiem i Denisovianem w ogóle miały miejsce, to były raczej incydentalne niż pospolite. Zastosowanie aDNA w tego rodzaju badaniach było możliwe dzięki rozwojowi i upowszechnieniu technik biologii molekularnej, szczególnie Łańcuchowej Reakcji Polimerazy oraz narzędzi bioinformatycznych pozwalających na coraz szybsze i dokładniejsze przyrównywanie sekwencji i gromadzenie danych. Analiza antycznego DNA nie jest jednak pracą łatwą i wspomniane techniki muszą poradzić sobie z całym szeregiem problemów będących efektem, przede wszystkim, czasu.
Fragmentacja
Fragmentacja materiału genetycznego jest podstawową cechą aDNA. Dochodzi do niej przede wszystkim w wyniku działania dwóch czynników- hydrolizy katalizowanej przez obecne w środowisku nukleazy oraz oddziaływania cząsteczki kwasu nukleinowego z promieniowaniem jonizującym, które generuje dwuniciowe pęknięcia w helisie DNA. Z tych dwóch czynników dominującą rolę odgrywa jednak hydroliza a o tym jak bardzo DNA zostanie pofragmentowany (w skrajnych przypadkach materiał genetyczny może być pocięty na fragmenty długości maksymalnie kilkudziesięciu par zasad, często jednak są to fragmenty 100-150pz, ryc.1) decydują warunki środowiskowe takie jak rodzaj otoczenia w jakim znajdował się materiał źródłowy, wilgotność czy temperatura. Przykładowo, materiał genetyczny znajdujący się w glebie bogatej w krzemionkę będzie bardziej odporny na hydrolizę niż DNA zawieszony w wodzie. Dzieje się tak, ponieważ DNA silnie wiąże się do krzemionki przez co staje się niedostępny dla enzymów hydrolitycznych, efekt ten jest wzmacniany w obecności soli chaotropowych rozbijających wiązania wodorowe w tym przypadku pomiędzy cząsteczkami wody a złożem krzemionki dzięki czemu zwiększa się powinowactwo cząsteczek DNA do złoża. Zjawisko to wykorzystuje się również podczas izolacji DNA z wykorzystaniem mikrokolum, gdzie cząsteczki DNA wiążą się do złoża krzemionkowego pełniącego rolę filtra wyłapującego DNA. Sole chaotropowe występują naturalnie w środowisku a przykładem takiego związku jest pospolity chlorek magnezu. W podobny sposób DNA jest chronione przed hydrolizą kiedy tworzy wiązania z kwasami humusowymi obecnymi np. w odchodach zwierząt.
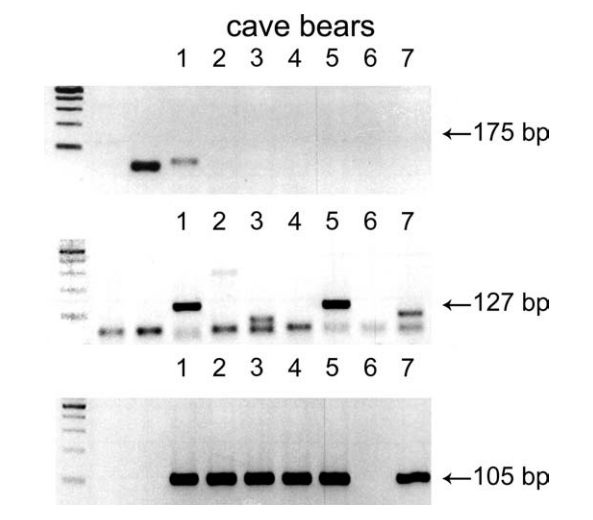
Ryc.1. Rozdział elektroforetyczny fragmentów aDNA jakie udało się uzyskać podczas amplifikacji materiału genetycznego Niedźwiedzia jaskiniowego (Ursus spelaeus) wymarłego około 28 tysięcy lat temu. Źródło: [1]
Połączenia krzyżowe
Kolejnym uszkodzeniem często występującym w antycznym materiale genetycznym są połączenia krzyżowe (ryc.2) w obrębie jednej cząsteczki lub pomiędzy różnymi cząsteczkami DNA. Powstają one między innymi w wyniku działania reaktywnych form tlenu, które doprowadzają do utleniania zasad azotowych i reszt cukrowych w kwasach nukleinowych co z kolei umożliwia powstawanie wiązań pomiędzy nimi. Tego typu uszkodzenia są o tyle problematyczne, że unimożliwiają przeprowadzenie reakcji PCR poprzez hamowanie aktywności polimerazy lub całkowite unimożliwienie jej przyłączenia, co ważne, sama obecność utlenionych form zasad może stanowić czynnik hamujący aktywność polimerazy
Ryc.2. Obraz z mikroskopu elektronowego przedstawiający połączone krzyżowo DNA wyizolowane z fragmentu tkanki wątroby pobranej od egipskiego kapłana Nekht-Ankh. Materiał ten pochodził sprzed 4 tysięcy lat. [Autor zdjęcia: Svante Pääbo, Uniwersytet Maxa Plancka]
Deaminacja
Deaminacja DNA jest reakcją chemiczną zachodzącą zarówno spontanicznie jak i z udziałem specyficznych enzymów hydrolitycznych polegającą na usunięciu z zasady azotowej grupy aminowej. Proces ten prowadzi do wbudowania nieprawidłowych nukleotydów w nowo syntetyzowanej nici DNA podczas amplifikacji ponieważ obecność grupy aminowej w danym miejscu jest tym co odróżnia np. cytozynę od uracylu (Rys.3) czy adeninę od hipoksantyny (która jest komplementarna do cytozyny). W efekcie można otrzymać nieprawidłowe wyniki sekwencjonowania, w których zostaną wykazane sekwencje zmienione już po śmierci organizmu, co stanowi problem przy analizach filogenetycznych.
Depurynacja i depirymidynacja
Do obu tych uszkodzeń dochodzi poprzez ten sam mechanizm. W wyniku hydrolizy wiązanie β-N-glikozydowe pomiędzy zasadą azotową a rybozą lub deoksyrybozą zostaje zerwane. Zasada zostaje uwolniona pozostawiając w nici DNA puste miejsce, są to tak zwane miejsca apurynowe w przypadku adeniny (Rys. 4) i guaniny lub apirymidynowe w przypadku cytozyny oraz tyminy. Takie pozbawione zasad azotowych pozycje w sekwencji DNA są problemem z dwóch powodów. Po pierwsze następuje utrata informacji o pełnej sekwencji analizowanego fragmentu i co więcej może to prowadzić do wbudowania przez polimerazę losowego nukleotydu podczas amplifikacji. Powstawanie miejsc pozbawionych zasad silnie zależy zarówno od temperatury otoczenia, w którym znajduje się materiał źródłowy oraz od samej sekwencji DNA.
Kontaminacja
Antyczny DNA, zwłaszcza ten izolowany z prób środowiskowych takich jak osady czy pokłady lodowcowe może być i praktycznie zawsze jest silnie zanieczyszczony zarówno inhibitorami hamującymi lub uniemożliwiającymi reakcję PCR (w ten sposób działają korzystne dla trwałości DNA w środowisku kwasy humusowe) jak i całą gamą heterogennego DNA, to znaczy materiału genetycznego pochodzącego z różnych źródeł zarówno antycznych jak i współczesnych . Takie zanieczyszczenie heterogennym DNA jest istotnym problemem w przypadku badań, które nie mają na celu analizy metagenomu środowiskowego, a skupiają się na konkretnym gatunku lub innym, większym, ale wciąż konkretnym taksonie.
Metodyka pracy z aDNA
Pomimo wspomnianych cech jakimi charakteryzuje się antyczny DNA zastosowanie odpowiednich technik a także specyficznej organizacji laboratorium pozwala na rzetelną analizę materiału i wyciągnięcie z opracowanych danych tak samo rzetelnych wniosków. Część uszkodzeń DNA jest w warunkach laboratoryjnych odwracalna poprzez zastosowanie odpowiednich odczynników takich jak PTB (bromek N-fenyloacylotiazoliowy), który rozbija wiązania krzyżowe w DNA. Innym wykorzystywanym odczynnikiem jest glikozylaza uracylu wycinająca powstały w wyniku deaminacji uracyl.
Problem fragmentacji materiału genetycznego rozwiązuje się poprzez amplifikację nachodzących na siebie odcinków. Z uwagi na potencjalną ilość uszkodzeń w badanej próbce aDNA analiza fragmentów jest nawet bardziej korzystna niż analiza jednej lub kilku długich odcinków. Jest tak, ponieważ uzyskanie poprzez PCR dużej liczby kopii krótkich odcinków da nam dodatkową korzyść przy sekwencjonowaniu metodą NGS, mianowicie wykorzystując tą technikę jesteśmy w stanie na podstawie wielu odczytów wykonanych na uzyskanych amplikonach stworzyć tak zwaną sekwencję konsensusową. Jest to taka sekwencja, którą uzyskuje się poprzez przyrównanie do siebie odczytów sekwenatora, obliczenie częstości występowania danego nukleotydu w każdej pozycji wszystkich odczytów i wyróżnienie takiej sekwencji, która charakteryzuje się największą powtarzalnością. Pozwala to dodatkowo ominąć kwestię ewentualnej kontaminacji innym DNA, jeżeli te zanieczyszczenia występują w znacznej mniejszości w stosunku do DNA będącego obiektem badań.
Kwestią, która wymaga najwięcej pracy i precyzji, a także nakładu finansowego jest zapobieganie kontaminacji współczesnym DNA. Laboratorium zajmujące się badaniem DNA antycznego musi być koniecznie oddzielone od laboratoriów, tak zwanych, post-PCR. Zaleca się wręcz, żeby pomieszczenie do pracy z aDNA znajdowało się w innym budynku niż laboratoria, w których znajdują się innego typu materiały badawcze lub, żeby miało osobne wejście. Wewnątrz placówki należy wydzielić miejsca, dla każdego z etapów analizy aDNA w taki sposób, aby pomieszczenie przeznaczone do analizy próbek post-PCR było dostępne tylko z części, w której PCR się prowadzi, a to z kolei powinno być dostępne tylko z pomieszczena przeznaczonego do izolacji. Ponad to, w każdym pomieszczeniu należy zapewnić osobne odczynniki. Niezbędna jest też śluza, oświetlenie UV a także filtry powietrza zapobiegające kontaminacji DNA przynoszonym spoza placówki. Ważne jest także ubranie ochronne zapobiegające przenoszeniu DNA w czasie korzystania z narzędzi czy odczynników.

Ubiór ochronny i wyposażenie jednego z pomieszczeń w laboratorium antycznego DNA. Źródło [7]
Literatura:
- Svante Pääbo et al. Genetic analyses from ancient DNA. Annu. Rev. Genet. 2004. 38: 645-79
- Pedersen MW et al. Ancient and modern environmental DNA. Phil. Trans. R. B. 2015. 370:20130383
- Yun-Jeong Kim, David M. Wilson. Overview of base excision repair biochemistry. Curr. Mol.Pharmacol. 2012. 5(1): 3-13
- An R. et al. Non-enzymatic depurination of nucleic acids: factors and mechanisms. PloS ONE. 2014. 9(12)
- Ermanno Rizzi et al. Ancient DNA studies: new perspectives on old samples. Genet Sel Evol. 2012. 44(1): 21
- Michael Knapp et al. Setting the stage- Building and working in an ancient DNA laboratory. Annals of Anatomy. 2012. 194: 3-6.
- http://science.psu.edu/alert/photos/miscphotos/SchusterMiller/AncientDNAlab1_Lrg.jpg/ view. Dostęp 05.08.2018, godz. 16:25
- Jason A. Hodgson, Todd R. Disotell. No evidence of a Neanderthal contribution to modern human diversity. Genome Biology. 2008. 9: 206.
- Kwang Hyun Ko. Hominin interbreeding and the evolution of human variation. J. Biol. Res. 2016. 23: 17

