Autor: Anna Kurcek
Transkrypcja to proces przepisywania informacji genetycznej zawartej w DNA na cząsteczkę RNA. Jej podstawą jest reguła komplementarności (łac. cemplementum – dopełnienie, uzupełnienie) obowiązująca podczas parowania się zasad azotowych. Dzięki niej sekwencja nukleotydów w nowo-syntetyzowanym RNA jest jednoznacznie określona przez kolejność zasad na matrycowej nici kwasu dezoksyrybonukleinowego.
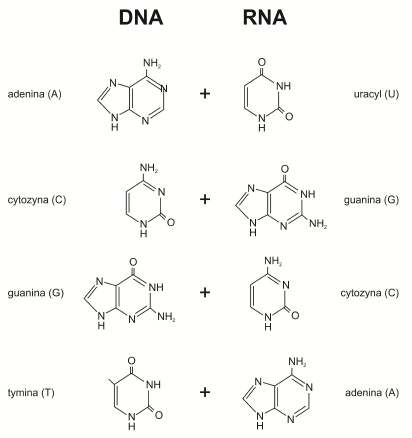
Rys. 1. Zasada komplementarności podczas transkrypcji
Transkrypcji podlega odcinek DNA znajdujący się między promotorem a terminatorem i nazywany jednostką traskrypcji. Nić DNA odczytywana jest w kierunku 3′ → 5′, natomiast cząsteczka RNA powstaje w kierunku 5′ → 3′. Proces katalizowany jest przez polimerazę RNA.
Etapy transkrypcji:
• Inicjacja – przyłączenie się polimerazy do promotora;
• Elongacja – wydłużenie nici RNA. Zachodzi w bąblach transkrypcyjnych, które poruszają się wzdłuż matrycy DNA. Substratami są trifosfonukleotydy, od których odłączane są dwie grupy fosforanowe dostarczając w ten sposób energii potrzebnej do wytworzenia połączenia między nukleotydami;
• Terminacja – zachodzi, gdy polimeraza RNA dotrze do sekwencji kończącej zwanej terminatorem.
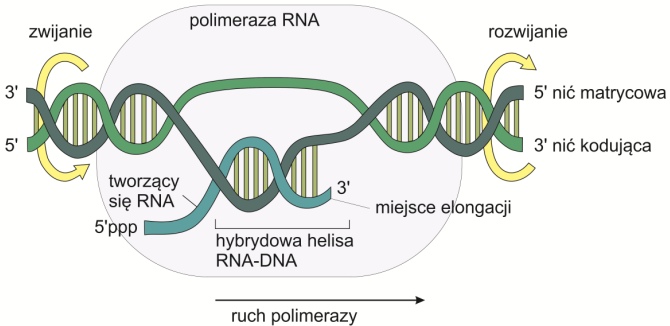
Rys. 2. Wydłużanie (elongacja) transkryptu RNA
Transkrypcja u Eucaryota jest o wiele bardziej złożona niż u Procaryota. Obecność otoczonego błoną jądra komórkowego warunkuje rozdzielenie transkrypcji i translacji w czasie i przestrzeni, co umożliwia wielostopniową regulację ekspresji genów. Powstały łańcuch jest poddawany dalszym obróbkom – dodanie kapu na końcu 5’, ogona Poli(A) na końcu 3’ oraz splicing. U Procaryota oba procesy są ze sobą powiązane – translacja rozpoczyna się, gdy transkrypt jest jeszcze syntetyzowany.
U bakterii występuje również tylko jeden rodzaj polimerazy RNA. Jest to kompleksowy enzym zbudowany jest z pięciu podjednostek. Dwie z nich – α wiążą białka regulatorowe, podjednostka β przyłącza substraty reakcji, natomiast β’ jest odpowiedzialna za wiązanie matrycy DNA. Razem tworzą one rdzeń enzymu, który przed syntezą łączy się z podjednostką σ odszukującą miejsce promotorowe na matrycy.
U Procaryota promotor położony jest w sąsiedztwie części kodującej genu, a liczba powstających transkryptów zależy od ilości cząsteczek polimerazy, które biorą w niej udział.
U Eucaryota promotor zbudowany jest z kilku części regulatorowych. Promotor rdzeniowy poprzedza miejsce startu transkrypcji i pełni rolę zbliżoną do swojego bakteryjnego odpowiednika. W jego obrębie znajdują się sekwencje TATA-box oraz CAAT-box. Wzmacniacze (ang. enhancers) i wyciszacze (ang. silencers) umiejscowione są poza obszarem rdzenia i często występują w znacznej odległości od odcinka kodującego dane białko.
Polimerazy u Eucaryota nie mogą same przyłączać się do promotora. Na ich powinowactwo wpływają niehistonowe białka pomocnicze, czyli tzw. czynniki transkrypcyjne (ang. transcription factors). Tworzą one kompleks transkrypcyjny, będący odpowiednikiem podjednostki σ u Procaryota.
Ten system regulacji transkrypcji nie dotyczy genów metabolizmu podstawowego (ang. housekeping genes), kodujących białka niezbędne do przebiegu procesów życiowych komórki.
Czynniki transkrypcyjne dzieli się na trzy typy:
• ogólne (ang. general factors) – inicjują syntezę RNA na wszystkich typach promotorów;
• wiodące (ang. upstream factors) – łączą się z sekwencjami położonymi przed miejscem startu transkrypcji;
• indukowalne (ang. inducible factors) – podobne do czynników wiodących, lecz syntetyzowane jedynie o określonym czasie i w określonych tkankach.
Białka regulatorowe posiadają charakterystyczne motywy sekwencyjne, które wyróżniają je spośród innych białek. Są to:
• palec cynkowy (ang. zinc finger domain) – posiada w swojej strukturze jeden lub więcej jonów Zn2+ stabilizujących domenę. Mogą być one otoczone przez dwie reszty cysteinowe i dwie reszty histydynowe (klasa C2H2) lub przez cztery reszty cysteinowe (klasa C4);
• zamek leucynowy (ang. leucine zipper) – to bogata w leucynę domena dimeryzacyjna, warunkująca hydrofobowe połączenie dwóch zwojów α-helisy;
• helisa-skręt-helisa (ang. helix-turn-helix; HTH) – wiąże się do głównego rowka DNA;
• helisa- pętla-helisa (ang. helix-loop-helix; HLH) – jest odpowiedzialna za dimeryzację.
U Eucaryota występują trzy rodzaje polimerazy RNA. Wszystkie są dużymi kompleksami białkowymi składającymi się z 8-12 podjednostek.
• polimeraza I RNA – niewrażliwa na α-amanitynę, umiejscowiona w jąderku. Prowadzi transkrypcję wszystkich rodzajów genów rRNA z wyjątkiem 5S rRNA. Do swojej aktywności wymaga obecności jonów Mg2+ oraz czynników transkrypcyjnych: UBF1 (ang. upstream binding factor) i UCF. Rozpoznaje promotory leżące w górę od miejsca startu transkrypcji;
• polimeraza II RNA – bardzo wrażliwa na α-amanitynę. Znajduje się w nukleoplazmie i jest odpowiedzialna za syntezę mRNA oraz większości snRNA. Wymaga obecności jonów Mg2+. Jej promotory zbudowane są z rdzenia (blok TATA – miejsce wiązania ogólnego czynnika TFIID) i dodatkowych sekwencji regulatorowych (ang. cis-acting elements) np. sekwencja CAAT, wzmacniacze (ang. enhancers), elementy RE (ang. response elements);
• polimeraza III RNA – wrażliwa na wysokie stężenia α-amanityny. Usytuowana w nukleoplazmie, przeprowadza transkrypcję genów 5S rRNA, tRNA oraz małych jądrowych RNA. Wymaga obecności jonów Mn2+ oraz ogólnych czynników transkrypcyjnych: TFIIIA, TFIIIB i TFIIIC tworzących kompleks pre-inicjacyjny. Wykorzystuje trzy typy promotorów, z których dwa są za miejscem startu transkrypcji (ang. downstream), antomiast trzeci posiada bliski i daleki promotor.
W regulacji ekspresji genów ważną rolę odgrywa również struktura chromatyny. Mocno upakowana heterochromatyna, w okresach wzmożonej transkrypcji może ulegać dekondensacji. W proces ten zaangażowany jest także histon H1, który w odmienny sposób wiąże się do obszarów aktywnych transkrypcyjnie i rejonów wyciszonych.
Literatura:
• Jerzy Bal „Biologia molekularna w medycynie. Elementy genetyki klinicznej”; Wydawnictwo Naukowe PWN; Warszawa 2001;
• Lubert Stryer „Biochemia, część V – Geny: replikacja i ekspresja”; Wydawnictwo Naukowe PWN, Warszawa 2003;
• http://arete.ibb.waw.pl/res/RadekTranskrypcja.html;
• Ewa Pyłka-Gutowska „Biologia – vademecum maturzysty”; Wydawnictwo „OŚWIATA”; Warszawa 2002r.

