Łańcuchowa reakcja polimerazy (ang. Polymerase Chain Reaction – PCR) jest to metoda pozwalająca na powielanie konkretnych odcinków DNA w warunkach laboratoryjnych. Została ona odkryta przez Kary’ego Mullisa w 1980 roku i od tego czasu opracowano już wiele jej ulepszeń oraz modyfikacji. Jedną z takich odmian jest tzw. real-time PCR, czyli inaczej PCR w czasie rzeczywistym, zwany również kinetyczną reakcją PCR (ang. kinetic PCR) lub ilościową reakcją PCR (ang. quantitive PCR lub quantitive fluorescent PCR). Nie należy jej jednak mylić z RT-PCR (ang. Reverse Transcription), w której jako matrycę stosuje się nici mRNA.
Real-time PCR umożliwia jednoczesne namnażanie DNA oraz monitorowanie ilości produktów powstających podczas kolejnych cykli (amplikonów). Po raz pierwszy została ona zaprezentowana przez zespół Higuchi’ego z Roche Molecular Systems and Chiron. Dzięki dodatkowi bromku etydyny (EtBr) oraz przeprowadzeniu reakcji pod ultrafioletem udało się im wzbudzić fluorescencję barwnika i tym samym zwizualizować oraz nagrać proces amplifikacji.
W skład mieszaniny reakcyjnej standardowej reakcji PCR wchodzą oligonukleotydowe primery, nukleotydy stanowiące substraty reakcji, bufor zapewniający właściwe środowisko, jony magnezu niezbędne do właściwego funkcjonowania enzymów oraz termostabilna polimeraza DNA – posiadająca zdolność do tworzenia nowych odcinków DNA z wykorzystaniem matrycy DNA oraz primerów. Powszechnie stosowane są Taq DNA polimeraza (Thermus aquaticus) oraz Pfu DNA polimeraza (Pyrococcus furiosus), która zapewnia wysoką wierność przy replikacji. Oba te enzymy są odporne na wysokie temperatury i dzięki temu mogą przetrwać kolejne cykle reakcji PCR. Po fazie namnażania podwójne nici DNA muszą bowiem ulec denaturacji w 95°C, aby w ten sposób umożliwić primerom przyłączenie się do jednoniciowych cząsteczek i tym samym rozpocząć nowy cykl. Teoretycznie, w każdym etapie liczba cząsteczek powinna ulec podwojeniu, jednak po fazie początkowej, charakteryzującej się logarytmicznym wzrostem następuje tzw. faza eksploatacyjna (faza plateau) będąca wynikiem zużywania się reagentów. Oznacza to, że liczba cząsteczek produktu może wzrosnąć tylko do pewnej wartości granicznej, a zjawisko to uniemożliwia określenie wyjściowej zawartości DNA na podstawie ilości produktu końcowego.
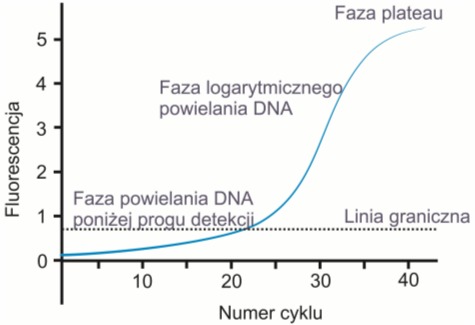
Rys. 1. Wykres fluorescencji w zależności od cyklu Real-time PCR
Dzięki zjawisku fluorescencji real- time PCR pozwala śledzić proces namnażania się DNA w czasie rzeczywistym i tym samym oznaczyć ilości produktu w fazie jego wykładniczego wzrostu. Jego wartość bezwzględną określa się dzięki sporządzeniu tzw. krzywej standardowej dla danego genu, to znaczy serii rozcieńczeń znanej ilości cząsteczek, służących za matryce w reakcji PCR. Wartość względna odnosi się do tzw. genów referencyjnych. Najczęściej są to geny metabolizmu podstawowego (ang. housekeeping genes), które charakteryzują się stałym i niekontrolowanym poziomem ekspresji w danej tkance (ang. „housekeeping genes”).
Real-time PCR umożliwia także dokonanie pomiarów początkowej ilości DNA, którą wyznacza się na podstawie cyklu, w którym sygnał fluorescencji osiągną wartość graniczną pozwalającą ma jego wykrycie (ang. treshold cycle Ct).
Dzięki tym udogodnieniom metoda real-time PCR szybko zyskała ogromną popularność. Pierwsze urządzenia i odczynniki niezbędne do jej przeprowadzania pojawiły się na rynku w 1996 roku i zostały wypuszczone przez firmę Applied Biosystem.
W skład typowego urządzenia wchodzą następujące elementy:
• źródło światła wzbudzające cząsteczkę reporterową – lampy halogenowe o szerokim spektrum emisji lub charakteryzujące się wąskim spektrum diody elektroluminescencyjne LED i laser;
• fotodetektor, czyli optyczne układy detekcji fluoresencji (soczewki, fotodiody, fotopowielacze); często są one zaopatrywane w dodatkowe filtry przepuszczające jedynie fale o pożądanej długości;
• termocykler – układ kontroli temperatury, który dzięki blokowi grzejnemu utrzymuje jednakowe warunki reakcji we wszystkich studzienkach. Próby mogą być również chłodzone i ogrzewane powietrzem;
• specjalne oprogramowanie, które umożliwia analizę uzyskanych danych. Wyniki mogą być przestawione graficznie w postaci wykresów – tzw. krzywej amplifikacji charakteryzującej kinetykę amplifikacji sekwencji docelowej oraz krzywej dysocjacji ujawniającej właściwości produktu końcowego.
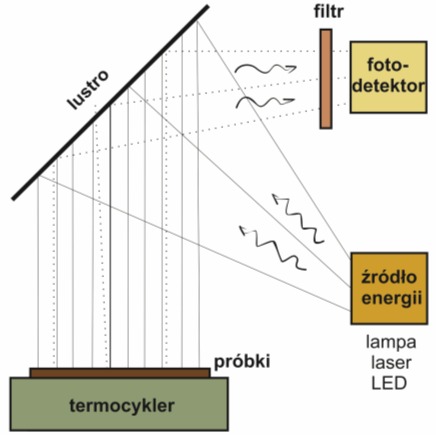
Rys. 2. Termocykler do Real-time PCR
Badane próby umieszcza się w termocyklerze, który kontroluje temperaturę podczas trwania poszczególnych cykli PCR. Próbki te są wystawione na działanie energii wzbudzającej, a powstająca fluorescencja mierzona jest dzięki obecności fotodetektora
Do monitorowania ilości powstających produktów stosuje się różne metody znakowania sond, primerów oraz amplikonów:
Metody niespecyficzne – wykorzystują barwniki wiążące się z DNA (np. SYBR Green I, BEBO, YOYO-1, TOTO-1)
Najpowszechniej stosowany jest SYBR Green I, który łącząc się do małego rowka podwójnej helisy dsDNA emituje 1000 razy większą fluorescencję niż wtedy, gdy jest zawieszony w roztworze. Im większa ilość związanego barwnika, tym silniejszy sygnał fluorescencji. Metoda ta nie wymaga kosztownego projektowania specyficznych starterów, jednak niesie ze sobą ryzyko, że barwniki połączą się np. ze sparowanymi primerami (ang. primer-dimer). Zagrożenie to zwiększa się wraz z liczbą przeprowadzonych cykli.
Specyficzność tych prób można sprawdzić za pomocą analizy temperatury topnienia, która jest cechą charakterystyczną dla każdego dwuniciowego odcinka DNA. Po zakończeniu PCR sporządza się tzw. krzywą topnienia, tzn. stopniowo podwyższa się temperaturę badanej mieszaniny. W momencie denaturacji sygnał fluorescencji gwałtownie się obniża, co jest obrazowane na wykresie. W przypadku wystąpienia niespecyficznych połączeń obserwuje się większą niż jeden liczbę pików.
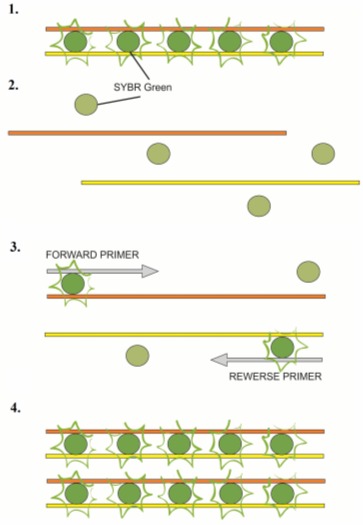
Rys. 3. Metoda z wykorzystaniem SYBR Green I
1. SYBR Green I połączony z dsDNA emituje sygnał fluorescencji; 2. denaturacja – SYBR Green I jest uwalniany i sygnał zanika; 3. polimeryzacja – powstawanie nowych produktów (ds.DNA); 4. zakończenie polimeryzacji – SYBR Green I łączy się ponownie z cząsteczkami dsDNA; im większa ilość związanego barwnika, tym silniejszy sygnał.
Metody specyficzne – z użyciem barwników specjalnie zaprojektowanych i zsyntetyzowanych dla danej sekwencji. Zasada ich działania opiera się na rezonansowym transferze energii fluorescencji (FRET) pomiędzy cząsteczką donora i wygaszacza.
• liniowe sondy oligonukleotydowe/ sondy sąsiadujące (ang. dual hybridisation probes/adjacent probes) – metoda z użyciem pary fluorescencyjnych sond oligonukleotydowych, emitujących sygnał jedynie wtedy, gdy zbliżają się do siebie w wyniku przyłączenia się do matrycy DNA.
Sonda poprzedzająca (ang. upstream) na końcu 3’posiada donorowy fluorochrom, natomiast druga sonda (ang. downstream) posiada akceptorowy fluorochrom na swoim końcu 5’. Energia wzbudzenia donora jest przekazywana na cząsteczkę akceptora, która dzięki temu emituje sygnał fluorescencji. Jednoczesne wykorzystanie dwóch sond warunkuje wysoką specyficzność.
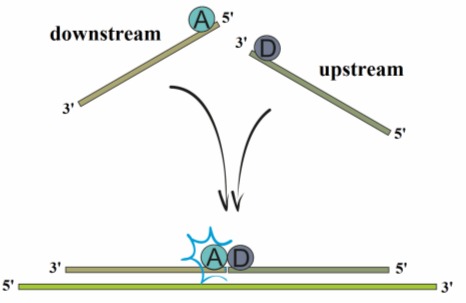
Rys. 4. Metoda z wykorzystaniem sond sąsiadujących. W sąsiedztwie donora (D) akceptor (A) wysyła sygnał fluorescencji
• sondy ulegające reakcji hydrolizy, czyli fluorescencja z wykorzystaniem aktywności 5’-nukleazowej polimerazy, (ang. 5′ Nuclease oligoprobes/ hydrolysis probes).
Przykładem mogą być sondy TaqMan. Pod względem strukturalnym są to fluorescencyjnie wyznakowane oligonukleotydy o specyficznej sekwencji. Jeden fluorofor nazywa się wygaszaczem (ang. quencher) drugi reporterem (ang. reporter). Gdy znajdują się one w bliskim sąsiedztwie, tzn. obie są przyłączone do tego samego oligonukleotydu, wygaszacz pochłania sygnał emitowany przez reporter. Podczas amplifikacji oligonukleotyd ulega rozdzieleniu dzięki aktywności 5’ egzonukleazowej polimerazy DNA. Reporter i wygaszacz zostają rozdzielone i w ten sposób zostaje uwolniony sygnał fluorescencji. Do najpowszechniejszych wygaszaczy należą: TAMARA, DABCYL, BHQ, natomiast wśród reporterów wyróżnia się FAM, VIC, NED oraz pochodne fluoresceiny (np. izotiocyjanian fluoresceiny, 6-karboksyfluoresceina). Tego typu sondy są równie precyzyjne co SYBR Green I, jednak cechuje je większa specyficzność. Pozwalają one również na wykrywanie ewentualnych mutacji punktowych zachodzących podczas powielania prób. Jeżeli w miejscu hybrydyzacji na amplikonach występują nieporządane zmiany, wtedy sondy są związane słabiej do cząsteczki DNA i oddysocjowują w niższej temperaturze. Zmiany te można zatem łatwo obserwować dzięki analizie krzywej dysocjacji.
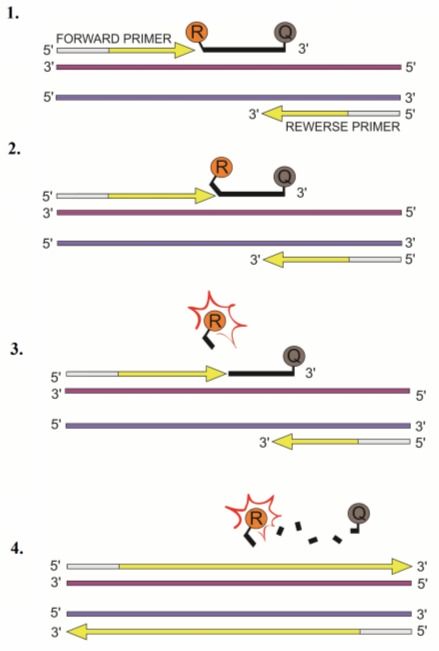
Rys. 5. Metoda z wykorzystaniem sond TaqMan
1. polimeryzacja –reporter (R) i wygaszacz (Q) są związane do oligonukleotydowej sondy; 2. przemieszczenie się nici – wygaszacz nadal pochłania sygnał emitowany przez reporter; 3. rozłam – wygaszacz odłącza się od sondy i oddala od reportera – sygnał staje się widoczny; 4. zakończenie polimeryzacji – odłączony reporter nadal emituje sygnał fluorescencji
• sondy o strukturze spinki do włosów (ang. hairpin oligoprobes) – dzięki obecności komplementarnych sekwencji wewnętrznych, ich drugorzędowa struktura przybiera charakterystyczną postać spinki do włosów, która jest niszczona podczas amplifikacji. Powoduje to rozdzielenie reportera i wygaszacza, a zatem wyzwolenie sygnału fluorescencji, który wcześniej był rozpraszany w postaci ciepła.
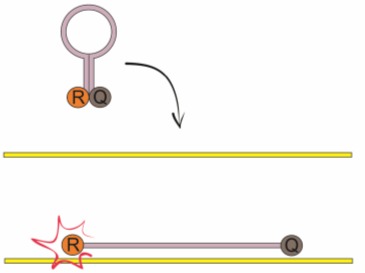
Rys. 6. Zasada działania sond o strukturze spinki do włosów. Po przyłączeniu się do komplementarnej sekwencji, reporter (R) i wygaszacz (Q) oddalają się od siebie, co umożliwia wykrycie sygnału fluorescencji
Tego typu sondy można podzielić na kilka rodzajów:
o markery molekularne (ang. molecular beacons) – w obecności komplementarnej matrycy DNA zmieniają one swoją konfigurację na otwartą, co powoduje rozdzielenie się reportera i wygaszacza;
o startery „sunrise” – czyli typu wschodzącego słońca (ang. sunrise primers) – to odmiana markerów molekularnych, wbudowywanych do wydłużającego się amplikonu. Wysoka specyficzność tych starterów powoduje, że muszą być one każdorazowo projektowane. Podczas ich stosowania występuje również ryzyko wystąpienia niespecyficznych sygnałów powstających w wyniku duplikacji sekwencji starterów;
o startery skorpion (ang. scorpion primers) – struktura spinki do włosów może być otwarta jedynie w obecności komplementarnej sekwencji amplikonu. Za właściwość tę odpowiada bloker połączony ze specyficznym starterem. Wyróżnia się dwa rodzaje sond skorpion – uni-sondy w których reporter, wygaszacz oraz bloker znajdują się na jednej cząsteczce oligonukleotydowej tworzącej strukturę spinki do włosów oraz bi-sondy, składające się z dwóch komplementarnych nici. Na jednaj z nich znajduje się reporter, bloker i starter, na drugiej zaś wygaszacz.
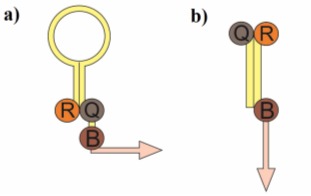
Rys. 7. Startery typu skorpion uni-sonda (a) i bi-sonda (b); Q – wygaszacz, R – reporter, B- bloker
Real-time PCR umożliwienie precyzyjne rozróżnienie i oznaczenie sekwencji kwasów nukleinowych nawet w bardzo małej próbce. Wysoka czułość tej metody, pozwala na wykrywanie nawet mniej niż pięciu kopi poszukiwanej sekwencji. Dzięki temu, że jest ona przeprowadzana w zamkniętym „pojemniku” nie wymaga żadnych dodatkowych manipulacji, które mogłyby zwiększyć zagrożenie występowania zanieczyszczeń zakłócających ostateczny wynik. Jest to również metoda stosunkowo szybka i wydajna.
Ze względu na te właściwości real-time PCR może być wykorzystywana do wykonywania analiz na obecność obcych fragmentów DNA (pochodzących z bakterii, wirusów, grzybów) w badanej próbie oraz ich ilościowego oznaczania. Określenie ilości wirusowego DNA jest pożytecznym wskaźnikiem rozwoju infekcji, relacji wirus-gospodarz oraz odpowiedzi na antywirusową terapię. Czynniki te odgrywają ważną rolę przy właściwym doborze metody leczenia m.in. u pacjentów po przeszczepach. Real-time PCR stosuje się także do oceny wektorów wirusowych jako narzędzi terapii genowej.
Jak każdy PCR odmiana real-time może zostać zakłócona przez pewne związki chemiczne występujące w badanych próbkach biologicznych, np. przez hemoglobinę i mocznik. Organiczne i fenolowe inhibitory zakłócają natomiast analizy mikrobiologiczne żywności. Tego typu niedogodnościom można zapobiegać używając odpornych polimeraz, jednak utrudnia to jej wykorzystywanie podczas niektórych badań klinicznych i kryminalistycznych. Największą przeszkodą podczas stosowania tej metody jest jednak czynnik ludzki i błędy powstające podczas analizowania uzyskanych wyników. Interpretacja danych jest jednym z najważniejszych i najbardziej narażonych na pomyłki etapów real-time PCR.
Zobacz również:
PCR
RT-PCR
Multipleks PCR
Nested PCR
MLPA PCR
Autor: Anna Kurcek
Literatura:
• Mark A. Valasek, Joyce J. Repa “The power of real-time PCR”; Adv Physiol Educ 29; 151-159, 2005;
• Monika Radwan , Dagmara Jonszta , Magdalena Kosz Vnenchak „Metoda PCR w czasie rzeczywistym ( Real-time PCR) – wyzwania i perspektywy”; Diagnosta Laboratoryjny, rok 6, nr. 2; sierpień 2008; str.10-17;
• Tomasz Romanowski, Aleksandra Markiewicz, Natalia Bednarz, Krzysztof Piotr Bielawski „Geny metabolizmu podstawowego jako geny referencyjne w ilościowym oznaczaniu ekspresji genów metodą real-time PCR”; Postepy Hig Med Dosw. (online), 2007; 61: 500-510;
• Ian M. Mackay, Katherine E. Arden, Andreas Nitsche “Real-time PCR in virology”; Nucleic Acids Res. 2002 March 15; 30(6): 1292–1305;
• http://www.appliedbiosystems.com.

