Operon to termin wprowadzony do genetyki w roku 1965 przez François Jacob’a i Jacques Monod’a. Mianem tym określa się jednostki tranksrypcyjne występujące u organizmów prokariotycznych w skład których wchodzi kilka genów kodujących zazwyczaj białka powiązane ze sobą funkcjonalnie. Dzięki obecności operonów możliwa jest regulacja ekspresji genów bakteryjnych, która pozwala mikroorganizmom odpowiadać na zmiany warunków zewnętrznych np. brak lub obecność substancji odżywczych. Operony możemy podzielić na indukowane i hamowane. Operony indukowane inaczej kataboliczne zawierają geny kodujące enzymy zaangażowane w procesach katabolicznych, ich ekspresja kontrolowana jest poprzez substrat. Produkcja enzymu następuje wyłącznie wtedy, gdy substrat jest obecny w środowisku. Przykładem operonu indukowanego jest operon laktozowy. Drugą grupę operonów stanowią operony hamowane (anaboliczne) kodujące głównie geny szlaków biosyntetycznych. Ekspresja tych genów jest kontrolowana przez produkt końcowy szlaku metabolicznego. Produkt ten może hamować ekspresję operonu lub też ją kontrolować poprzez atenuację. Przykładem tego rodzaju operonu anabolicznego jest operon tryptofanowy zawierający geny kodujące enzymy niezbędne w procesie syntezy tryptofanu.
Operon laktozowy
Operon laktozowy składa się z trzech leżących liniowo obok siebie genów kodujących enzymy zaangażowane w proces przyswajania laktozy przez bakterie Escherichia coli. Są to gen lac Z kodujący β-galaktozydazę, enzym hydrolizujący laktozę do glukozy i galaktozy, gen lac Y dla permeazy laktozowej – białka błonowego zaangażowanego w transport β-galaktozydów ze środowiska do wnętrza komórki oraz gen lac A kodujący transacetylazę β-galaktozydazową biorącą również udział w hydrolizie tego disacharydu. Geny te znajdują się w operonie pod jednym promotorem i są wspólnie transkrybowane, dając jedną cząsteczkę mRNA. W sąsiedztwie genów struktury znajduje się gen lac I kodujący represor zaangażowany w regulację ekspresji genów operonu laktozowego. W podstawowym modelu regulacji operonu laktozowego represor laktozowy spełniał rolę czynnika blokującego, który wiąże się z sekwencją DNA zwaną operatorem znajdującą się pomiędzy promotorem a początkiem genu lac Z i uniemożliwia polimerazie RNA dostęp do promotora. Dlatego w sytuacji braku laktozy transkrypcja trzech genów operonu jest zahamowana. W momencie pojawienia się w środowisku źródła laktozy bakteria może pobrać kilka jej cząsteczek i rozszczepić na glukozę i galaktozę. Produktem pośrednim w tej reakcji jest allolaktoza – izomer laktozy, który indukuje ekspresję operonu laktozowego poprzez połączenie się z represorem laktozowym. Allolaktoza wiążąc się z represorem zmienia jego konformację w taki sposób, że nie może on być dłużej związany z operatorem. Dzięki temu polimeraza RNA może związać się z promotorem i rozpocząć transkrypcję operonu. W warunkach pełnej indukcji w komórce znajduję się ok. 5000 cząsteczek każdego z białek. Po wyczerpaniu źródła laktozy, gdy brakuje allolaktozy, represor ponownie przyłącza się do operatora i wyłącza operon. Operon laktozowy podobnie jak inne operony kataboliczne podlegają równocześnie regulacji pozytywnej i negatywnej. Regulacja negatywna polega na przyłączeniu represora do operatora uniemożliwiając w ten sposób wiązanie polimerazy RNA i zachodzenie transkrypcji. W regulacji pozytywnej zwanej represją kataboliczną główną rolę odgrywa białko CAP będące aktywatorem katabolicznym. Białko to w obecności cAMP wiąże się z sekwencją DNA powyżej promotora, zwiększając w ten sposób powinowactwo polimerazy RNA do promotora a tym samym zwiększając aktywność transkrypcyjną operonu laktozowego. Mechanizm ten pozwala operonowi laktozowemu reagować na obecność glukozy w podłożu, która jest źródłem energii preferowanym przez komórkę. Dlatego też, jeśli zarówno glukoza jak i laktoza są obecne w otoczeniu komórki, poziom transkrypcji operonu laktozowego jest niski.
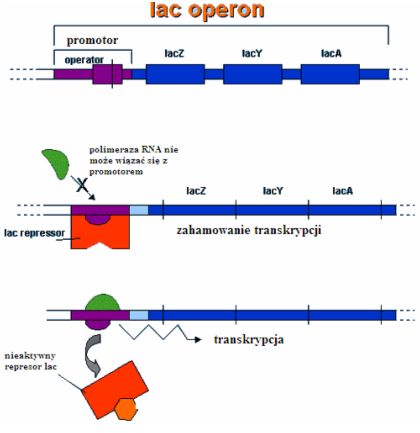
Rys.1 Operon laktozowy (3)
Operon tryptofanowy
Operon tryptofanowy składa się z 5 genów strukturalnych; trp E, trp D, trp C, trp B i trp A kodujących enzymy – syntetaza antranilowa, syntetaza indologlicerofosforanu, syntetaza tryptofanowi, zaangażowane w proces biosyntezy tryptofanu. Geny te, podobnie jak w operonie laktozowym posiadają jeden wspólny promotor i ulegają transkrypcji w postaci jednej cząsteczki RNA. Regulacja operonu tryptofanowego opiera się na systemie represor-operator podobnie jak w operonie laktozowym. Represja operonu zachodzi w obecności cząsteczki regulatorowej – tryptofanu, która jest produktem szlaku biochemicznego katalizowanego przez enzymy kodowane przez ten operon. Gdy tryptofan jest dostępny w otoczeniu komórki, operon jest wyłączony, ponieważ kompleks represor-tryptofan wiąże się do sekwencji operatora trp, uniemożliwiając w ten sposób związanie się polimerazy RNA z promotorem trp. W momencie braku tryptofanu, który działa jak korepresor, wspólnie z represorem blokując swoją syntezę. Represor nie może związać się z operatorem i zachodzi ekspresja operonu. Dodatkowo operon tryptofanowy zaopatrzony jest w dodatkowy mechanizm regulujący transkrypcję zwany atenuacją. Mechanizm ten opiera się na przedwczesnej terminacji transkrypcji, dzięki czemu pozwala na dostosowanie niektórych procesów biosyntezy do potrzeb komórki.
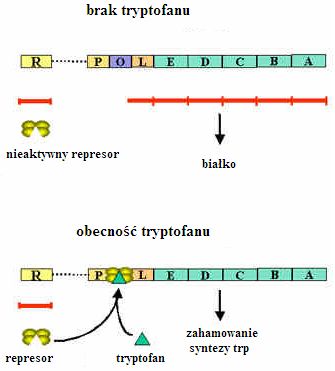
Rys.2 Operon tryptofanowy (4)
Autor: Beata Rola
Literatura:
1. Winter P.C., Hickey G.I., Fletcher H.L. 2009: Gnetyka. wyd. PWN Warszawa, 2009
2. Węgleński G. 2000. Genetyka molekularna. wyd. PWN Warszawa
3. http://lc-molecular.wikispaces.com/Worm+Feeding!
4. Mayer G.2010: Genetic regulatory mechanizm. Bacteriology, chapter 9

