Metylacja DNA jest to poreplikacyjna, enzymatyczna modyfikacja kwasów dezoksyrybonukleinowych, prowadząca do wyciszenia niektórych genów. Należy ona do tzw. zmian epigenetycznych, to znaczy takich, które mogą być przekazywane podczas podziałów komórkowych, pomimo, iż nie zależą od sekwencji DNA.
Ten rodzaj modyfikacji zachodzi głównie podczas fazy S cyklu komórkowego i obejmuje proces kowalencyjnego przyłączania grup metylowych (-CH3) do zasad azotowych nukleotydów.
Najczęstszymi produktami metylacji są:
• C5- metylocytozyna (m5C) – powstająca poprzez przyłączenie grupy metylowej do węgla w pozycji 5 pierścienia pirymidynowego cytozyny; występuje u Procaryota i Eucaryota;
• N4- metylocytozyna (m4C) – z grupą metylową przy azocie znajdującym się w pozycji 4 pierścienia cytozyny; występuje jedynie u Procaryota;
• N6- metyloadenina (m6A) – grupa metylowa przyłączona jest do azotu w pozycji 6 pierścienia purynowego adeniny; spotykana u Procaryota i u niższych organizmów eukariotycznych.
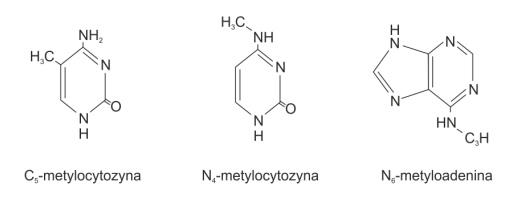
Rys. 1. Zmetydowane zasady azotowe
Miejsca metylacji nie są przypadkowe, a jej wzór jest cechą charakterystyczną dla danego gatunku, tkanki, a nawet typu komórki i ulega silnym zmianom w trakcie trwania rozwoju embrionalnego. Jej poziom zmniejsza się również wraz z wiekiem. W dorosłych, somatycznych tkankach, jest ona typowa dla rejonów CpG (dinukleotyd cytydyna-fosforan-guanozyna) i zachodzi symetrycznie na obu niciach. U kregowców około 70-80% dinukleotydów CpG ma grupę metylową przyłączoną do cytozyny. Metylacja niezwiązana z tymi regionami występuje w komórkach macierzystych embrionów.
Metylotransferazy
Proces metylacji katalizowany jest przez specjalną grupę enzymów – DNA-metylotransferaz (DNMT) adenino- lub cytozyno specyficznych. Donorem grup metylowych jest S-adenozylo-L-metionina (AdoMet), która w wyniku tej reakcji przekształca się w S-adenozylo-L-homocysteinę (AdoHcy).
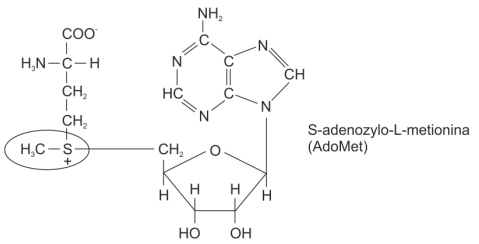
Rys. 2. S-adenozylo-L-metionina (AdoMet)
Do rodziny DNA-metylotransferaz zalicza się:
• DNMT1 – odpowiedzialną za przekazywanie stałego profilu metylacji podczas podziałów komórkowych; bierze ona udział w zachowawczej metylacji DNA, tzn. rozpoznaje hemimetylowane dinukleotydy CpG występujące na matrycowej lub potomnej nici DNA;
• DNMT2 – nie ma właściwości katalitycznych, jej rola nie jest dotychczas poznana; • DNMT3A i DNMT3B – odpowiadające za metylację de novo zachodzącą podczas embriogenezy i procesu różnicowania się komórek;
• DNMT3L – nie ma właściwości katalitycznych, ale zwiększa powinowactwo pozostałych metylotransferaz do DNA.
Metylacja DNA u Procaryota jest wynikiem naturalnej specyficzności metylotransferaz DNA. U ssaków enzymy te wymagają obecności specjalnych kofaktorów i adapterów, np. DNMT3L, UHRF1.
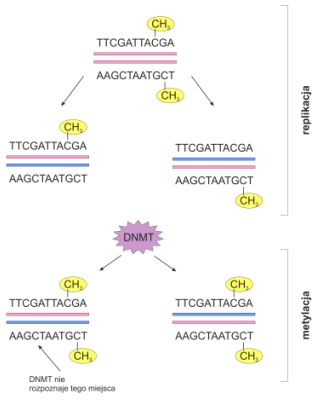
Rys. 3. Metylacja zachowawcza
Rola metylacji DNA
Jedynie niektóre geny ulegają stałej ekspresji. Są to tzw. housekeeping genes, warunkujące podstawowe funkcje życiowe. Transkrypcja pozostałych genów jest regulowana na drodze acetylacji i deacetylacji białek histonowych. Za pośrednictwem tych mechanizmów metylacja DNA może wpływać na strukturę chromatyny. Zmetylowane cytozyny są rozpoznawane przez grupę białek (MBDs) wiążących odpowiednie deacetylazy. Następująca w tym miejscu deacetylacja histonów powoduje utworzenie się zbitej, nieaktywnej struktury chromatyny.
Zmetylowanie nukleotydu uniemożliwia także przyłączenie się do niego czynników transkrypcyjnych.
Metylacja DNA jest więc kluczowym procesem warunkującym różnicowanie się komórek u wyższych organizmów. Umożliwia specjalizację komórek, przy ich jednoczesnym zaopatrzeniu w uniwersalną informację genetyczną i dzięki temu odgrywa bardzo ważną rolę w procesie embriogenezy. Zmniejsza ona również ekspresję genów wirusowych i innych szkodliwych elementów włączonych do genomu gospodarza.
Metylacja i imprinting rodzicielski
czyli inaczej piętnem genomowym lub piętnem rodzicielskim.
W nowo powstałym organizmie, ekspresji ulega tylko jeden allel danego genu (tzn. pochodzący od ojca lub od matki). Piętno to ustala się podczas gametogenezy i nie ulega zmianom w trakcie rozwoju osobniczego. Mechanizm jego powstawania polega na metylacji DNA podczas gametogenezy, w wyniku której geny tracą swoją aktywność. Zaburzenia tego procesu mogą powodować powstawanie chorób takich, jak np. zespołu Angelmana (AS) , Pradera-Williego (PWS), Beckwitha-Wiedemana (BWS) oraz transformacji nowotworowych. Metylacja jest zazwyczaj usuwana podczas formowania się zygoty i przywrócona w trakcie trwania podziałów komórkowych następujących podczas rozwoju organizmu.
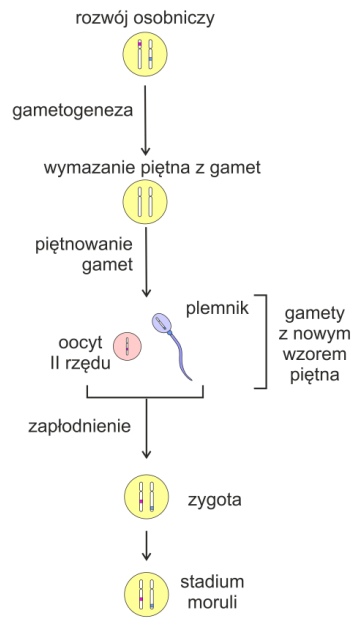
Rys. 4. Piętnowanie gamet – genów matki (kolor czerwony) i ojca (kolor niebieski)
Inaktywacja chromosomu X
zachodząca w komórkach samic ssaków łożyskowych.
Oba chromosomy X są aktywne jedynie w stadium wczesnego zarodka. W komórkach płodu poziom ekspresji genów zlokalizowanych na allosomach jest wyrównywany przez losową inaktywację jednego chromosomu. Proces ten zachodzi m. in. na drodze metylacji wysp CpG, a powstałe zmiany są przekazywane komórkom potomnym podczas podziałów mitotycznych.
Metylacja i nowotwory
Inicjacja procesu nowotworzenia może wynikać nie tylko z nagromadzenia się w komórce zmian genetycznych, ale również epigenetycznych, tzn. uniemożliwiających prawidłową ekspresję genów przy jednoczesnym zachowaniu ich niezmienionej sekwencji nukleotydowej. Taka komórka przestaje podlegać mechanizmom regulującym jej wzrost i różnicowanie. Obserwuje się w niej zmiany metylacji polegające na przyłączaniu grup metylowych de novo lub obniżeniu poziomu metylacji genomu.
Typy zmian:
o hipermetylacja – zachodzi w obrębie wysp CpG i powoduje wyciszenie genów supresorowych lub genów odpowiedzialnych za naprawę DNA, dotyczy głównie chromosomów 3p, 11p, 17p;
o hipometylacja zmetylowanych genów (globalna i lokalna) – demetylacja regionów promotorowych powoduje niestabilność chromosomów, aktywacja protoonkogenów, aktywacja transpozonów;
o tranzycja: 5-metylocytozyna (m5C) do tyminy – spontaniczna deaminacja, podczas której powstaje tymina – nieusuwana przez mechanizmy naprawcze. Deaminacja cytozyny powoduje powstawanie Uracylu, który może być usuwany z nici DNA. Metylacja adeniny i m6A nie jest mutagennym czynnikiem, gdyż daje ten sam produkt – hipoksantynę;
o metylacja „non-CpG” w komórkach nowotworowych – metylacja regionów niezwiązanych z CpG, najprawdopodobniej katalizowana przez DNMT3, DNMT3A i DNMT3B.
Związki powodujące takie zmiany nazywa się niegenotoksycznymi kancerogenami (NGCs).
Wykrywanie metylacji
Odstępstwa w metylacji są cechą charakterystyczną dla niektórych chorób, np. nowotworów, zaburzeń neurologicznych lub psychicznych. Zmetylowana cysteina może być zatem wykorzystywana jako biomarker. Istnieje wiele metod wykrywania metylacji DNA: (REP – PCR; MS-PCR; BSSCP; BGS). Ich dobór powinien być warunkowany ilością oraz jakością materiału biologicznego, z którego wyizolowano DNA.
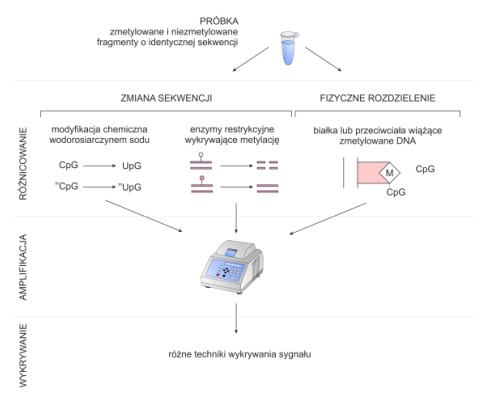
Rys. 5. Wykrywanie metylacji DNA – schemat ogólny
Autor: Anna Kurcek
Literatura: 1. Marcin Łukasik, Jolanta Karmalska, Mirosław M. Szutowski, Jacek Łukaszkiewicz „Wpływ metylacji DNA na funkcjonowanie genomu”, BIULETYN Wydziału Farmaceutycznego Warszawskiego Uniwersytetu Medycznego 2; 13-18; 2009;
2. Marta Olszewska, Maciej Kurisz „Metylacja i jej rola regulacyjna wobec rodzicielskiego piętna genomowego” Postępy Higieny i Medycyny Doświadczalnej 64; 642-649; 2010;
3. Grażyna Kostka, Katarzyna Urbanek „Metylacja DNA – alternatywny mechanizm chemicznej kancerogenezy”, ROCZN. PZH, 2005, 56(1), 1-14;
4. Victor V Levenson „DNA methylation as a universal biomarker”; Expert Rev Mol Diagn. 2010 May; 10(4): 481–488;
5. Steen K. T. Ooi, Anne H. O’Donnell, and Timothy H. Bestor “Mammalian cytosine methylation at a glance”; J Cell Sci. 2009 August 15; 122(16): 2787–2791.

